基于U-Net的自动分割方法对乳腺癌危及器官的自动勾画
引言
随着放射治疗技术的发展,其已成为肿瘤治疗的重要手段之一。据估计,美国每年有多达一半的癌症患者将接受放射治疗[1]。在肿瘤放射治疗的过程中,危及器官(Organs at Risks,OARs)及靶区的准确勾画是重要的步骤之一。通常都由医师对相应的CT图像进行手动勾画OARs及靶区,而手动勾画这些结构不仅耗时费力,而且勾画结果更依赖于医生所掌握的解剖学和肿瘤学知识,主观性较强,一致性和规范性较差。
近年来,有越来越多的研究致力于OARs自动勾画。当前已有多种类型的自动勾画技术,其中包括基于图谱的自动勾画技术[2-3],基于机器学习的自动勾画技术和基于神经网络的自动勾画技术[4-6]。基于图谱的自动勾画技术是通过目标CT图像与图谱库内的参考图像进行形变配准后根据图谱库内图像所勾画的结构对目标图像进行勾画。其缺点就是难以处理患者之间的解剖学差异[7],且建立图谱库时需花较长的时间。目前应用最多的是基于卷积神经网络(Convolutional Neural Networks,CNNs)的自动分割技术。自Ronneberger等[8]第一次提出U-Net用于生物医学图像分割开始,U-Net便被广泛应用于医学图像的自动分割。这得益于U-Net可以对较少的数据进行训练,勾画准确性较高,速度快等特点[8-9]。近年来,有许多研究提出一些结构类似于U-Net的一些变体用于图像的自动分割[10-12]。本研究意在训练和评价一种基于U-Net的乳腺OARs的自动分割方法。
1 材料与方法
1.1 实验数据
选取南京医科大学第一附属医院2018年4月至2019年11月的140例早期乳腺肿瘤保乳术后放疗的病例,其中左侧保乳术后及右侧保乳术后各70例,所有病例皆无锁骨以上靶区。定位时均采用仰卧位,双手上举置于托架上来固定患者体位。所有病例的图像采集均使用西门子CT模拟定位系统进行扫描,扫描层厚5 mm,层间距5 mm。将采集的CT图像通过DICOM传至MIM Maestro软件,由同一位医师在此软件上对所有病例进行勾画,并由高年资医师进行二次确认。所有病例所勾画的OARs为双肺,心脏及健侧乳腺(包括左侧正常乳腺及右侧正常乳腺),勾画标准参考RTOG 1106勾画指南[13]。
1.2 自动勾画方式
1.2.1 基于U-Net的自动勾画方法
使用120例已勾画好OARs的乳腺癌病例,其中左侧及右侧各60例。将病例标记为左右侧后用于训练自动勾画模型,训练过程如图1所示,首先将图像进行Dicom预处理,生成原始图像及标签图像,然后对原始图像进行增强及标准化后输入U形网络结构(图2),最后采用Dice损失函数对网络的预测结果和标签图像进行损失计算并反向传播。实验坏境:CPU:Intel(R)Xeon(R) CPU E5-2678v3@2.50GHz;GPU:NVIDIA TITAN RTX 24GB。
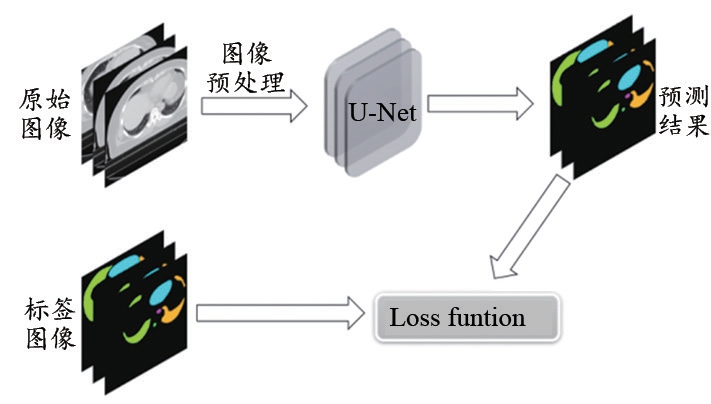
图1 训练流程
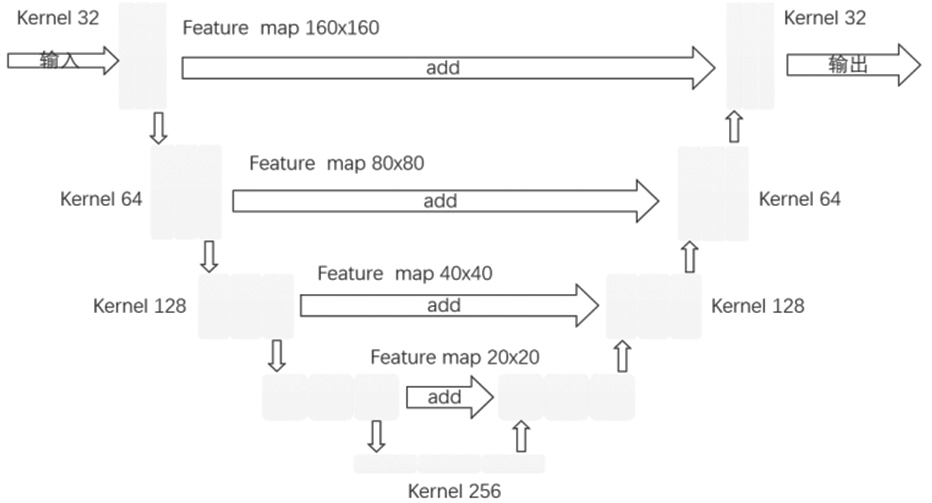
图2 U形网络结构
1.2.2 基于atlas的自动勾画方法
使用120例已勾画好OARs的乳腺癌病例,其中左、右侧乳腺癌各60例。将病例逐个导入MIM Maestro软件,数据导入后,标记每个病例的肿瘤左右轮廓并保存为atlas对象,进而在软件上创建自动勾画图谱库。
1.2.3 自动勾画的测试
使用20例乳腺癌病例的CT图像输入经训练好的基于U-Net及基于atlas的自动勾画模型进行勾画,将基于U-Net的自动勾画模型所勾画完成的数据导入MIM Maestro软件,在MIM Maestro软件上分别计算基于U-Net的自动勾画方法与手动勾画方法之间及基于atlas的自动勾画方法与手动勾画方法之间的各个OARs 的DSC值以及MDA值。
1.3 评估方法
采用戴斯相似系数(Dice Similarity Coefficient,DSC)及平均最小距离(Mean Distance to Agreement,MDA)对自动勾画的精度进行评估。
(1)DSC: 用于评价自动勾画和手动勾画之间的体积重叠性[14-15],公式(1)为:
其中,Va代表自动勾画轮廓的体积,Vm代表手动勾画轮廓的体积。DSC的大小在0至1之间,其数值越接近1表示相似度越高,则表明自动勾画的勾画精度越高。
(2)MDA: 用来描述两个集合之间的距离,具体含义是自动勾画轮廓集合中的所有点移动到与手动勾画轮廓集合完全重叠一致所需的平均最小距离[16-17],公式(2)为:
A代表自动勾画轮廓的点集,B代表手动勾画轮廓的点集,a∈A,a∈B,当MDA数值越接近于0,表明两个勾画轮廓集合之间的差别越小,则自动勾画精度越高。
1.4 统计学分析
使用SPSS 23.0软件对数据进行统计学分析。双侧肺,健侧乳腺,心脏的DSC值及MDA值用均数±标准差(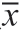 ±s)表示,对基于U-Net的自动勾画方法及基于atlas的自动勾画方法经行配对t检验,P值小于0.05则认为具有统计学差异。
±s)表示,对基于U-Net的自动勾画方法及基于atlas的自动勾画方法经行配对t检验,P值小于0.05则认为具有统计学差异。
2 结果
2.1 基于U-Net的自动勾画方法的勾画准确性评价
如表1所示,基于U-Net的自动勾模型所勾画的OARs,除了左侧正常乳腺DSCu均值为0.896,其余OARs的DSCu均值均大于0.9。其中双肺DSCu均值均大于0.972且MDA均值均小于0.62;健侧乳腺DSCu均值均大于0.89且MDAu均值均小于1.68;心脏DSCu均值为0.949,MDAu均值为1.206。图3a及图3b为基于U-Net的自动勾画方法对左、右侧乳腺癌OARs的勾画,如图所示,自动勾画方法所勾画的轮廓与医生手动勾画轮廓基本一致,在正常乳腺的起始及结束层面的勾画还有所欠缺,还需进一步修正。
表1 基于U-Net的自动勾画方法的OARs勾画准确度的评估参数(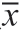 ±s,mm)
±s,mm)

OARs DSCu MDAu右侧正常乳腺 0.906±0.029 1.374±0.505左侧正常乳腺 0.896±0.012 1.673±0.525左肺 0.972±0.004 0.594±0.058右肺 0.975±0.004 0.619±0.073心脏 0.949±0.010 1.206±0.249
2.2 两种自动勾画方法的比较
如表2所示,双侧肺,左、右侧正常乳腺及心脏的DSCu均值均大于DSCa均值,且MDAu均值均小于MDAa均值。其中左、右侧正常乳腺的DSCu均值与DSCa均值差值最大,双侧肺的DSCu均值与DSCa均值差值最小;左、右侧正常乳腺的MDAu均值与MDAa均值差值最大,双侧肺的MDAu均值与MDAa均值差值最小。如图4所示,可以清楚的看到在双侧肺DSC折线图中,除了个别点之外,由基于U-Net的自动勾画模型勾画的双侧肺的DSC折线均位于由基于atlas的自动方法勾画的双侧肺的DSC折线之上,在MDA折线图中,除了个别点之外,由基于U-Net的自动勾画模型勾画的双侧肺的MDA折线均位于由基于atlas的自动方法勾画的双侧肺的MDA折线之下;心脏的DSC及MDA折线图分布与双肺相似,除个别点之外,在DSC折线图中,基于U-Net的自动勾画模型的勾画的心脏的DSC折线均位于由基于atlas的自动方法勾画的心脏的DSC折线之上,在MDA折线图中,基于U-Net的自动勾画模型勾画的心脏的MDA折线均位于由基于atlas的自动方法勾画的心脏的MDA折线之下;在左、右健侧乳腺中,则可以看到在DSC折线图中,由基于U-Net的自动勾画模型勾画的健侧乳腺的DSC折线均位于由基于atlas的自动方法勾画的健侧乳腺的DSC折线之上,在MDA折线图中,除个别点之外,由基于U-Net的自动勾画模型勾画的健侧乳腺的MDA折线均位于由基于atlas的自动方法勾画的健侧乳腺的MDA折线之下。使用配对t检验对两种勾画方法对乳腺癌OARs的勾画结果进行统计学分析,结果显示,双侧肺,左、右侧正常乳腺及心脏的DSC差异具有统计学意义(P<0.05)。左侧肺,左、右侧正常乳腺及心脏的MDA差异具有统计学差异(P<0.05),详见表2。如图3所示,基于U-Net的自动勾画方法的勾画轮廓与手动勾画轮廓重合性较基于atlas的自动勾画方法的好,且较其他OARs,健侧乳腺的勾画差异更为显著。

图3 两种自动勾画方法的OARs勾画结果示意图
注:绿色为手动勾画结果,红色为基于U-Net的自动勾画结果,蓝色为基于atlas的自动勾画结果。a和b为基于U-Net的自动勾画方法及手动勾画方法对右侧及左侧乳腺癌OARs勾画的比较;c和d为基于atlas的自动勾画方法及手动勾画方法对右侧及左侧乳腺癌OARs勾画的比较。
表2 两种自动勾画方法OARs勾画结果的比较( ±s)
±s)
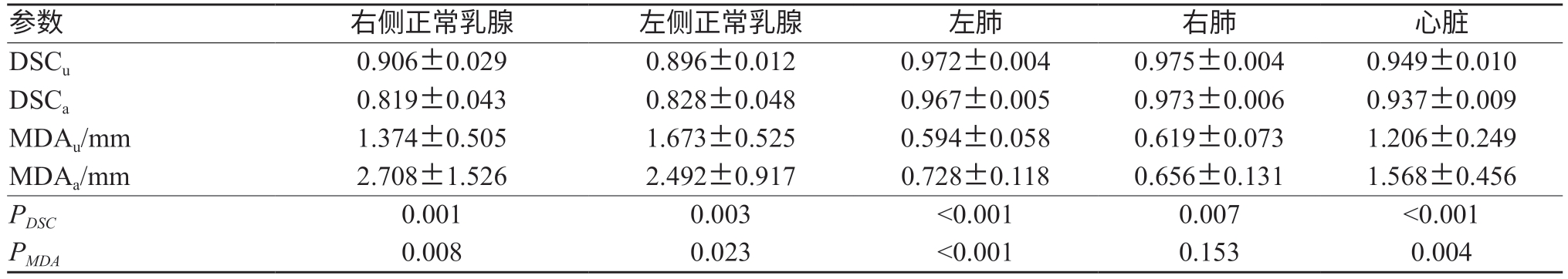
注:DSCu及MDAu表示基于U-Net的自动勾画方法的勾画结果评价参数,DSCa及MDAa表示基于atlas的自动勾画技术的的勾画结果评价参数。
参数 右侧正常乳腺 左侧正常乳腺 左肺 右肺 心脏DSCu 0.906±0.029 0.896±0.012 0.972±0.004 0.975±0.004 0.949±0.010 DSCa 0.819±0.043 0.828±0.048 0.967±0.005 0.973±0.006 0.937±0.009 MDAu/mm 1.374±0.505 1.673±0.525 0.594±0.058 0.619±0.073 1.206±0.249 MDAa/mm 2.708±1.526 2.492±0.917 0.728±0.118 0.656±0.131 1.568±0.456 PDSC 0.001 0.003 <0.001 0.007 <0.001 PMDA 0.008 0.023 <0.001 0.153 0.004
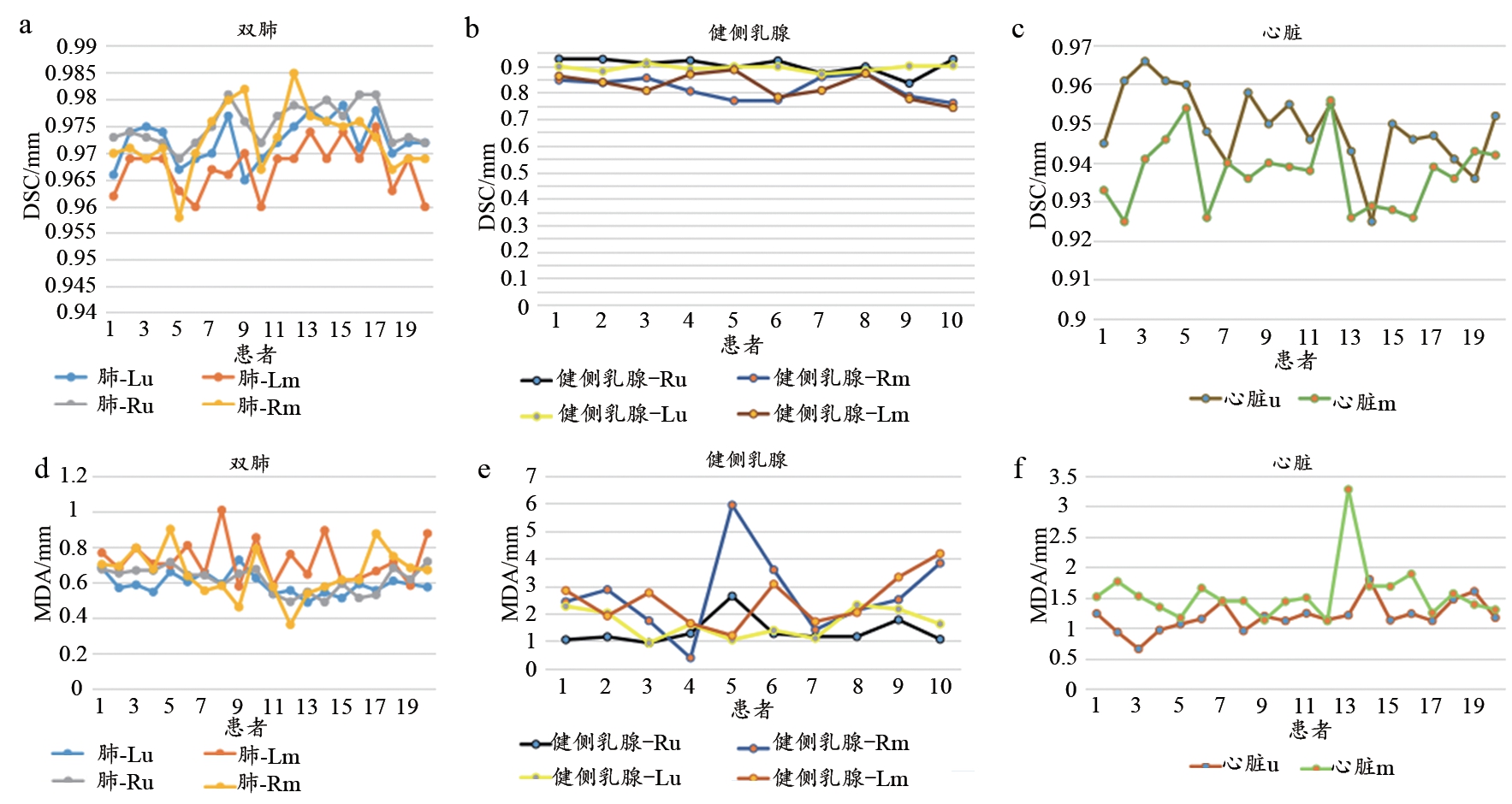
图4 两种自动勾画方法的OARs勾画结果折线图
注:a~c为两种勾画方法勾画的双肺、健侧乳腺、心脏的DSC的折线分布图,d~f为两种勾画方法勾画的双肺、健侧乳腺、心脏的MDA的折线分布图。肺-Lu、肺-Ru、心脏u、健侧乳腺-Lu、健侧乳腺-Ru均由基于U-Net的自动勾画方法所勾画;肺-Lm、肺-Rm、心脏m、健侧乳腺-Lm、健侧乳腺-Rm均由基于atlas的自动勾画方法所勾画。
3 讨论
随着精准放疗时代的到来,提高OARs勾画的准确性和一致性成为放射治疗的关键步骤之一。通常手动勾画OARs需要数十分钟而自动分割技术只需数十秒,因此图像的自动分割技术有利于加快放疗进程从而防止肿瘤因为定位后的长时等待导致的进展。同时其也有利于促进自适应放疗技术的发展,可缩短每日用于勾画OARs所需消耗的时间[12]。本研究训练了一种基于U-Net的自动分割方法用于早期乳腺癌OARs的自动分割。本研究训练的自动分割方法对双侧肺的勾画效果较好(DSC均大于0.972),在临床应用过程中医师基本不需要调整或只需要进行微调即可。左、右侧正常乳腺及心脏的DSCu值分别为0.896,0.906及0.949。Zijdenbos等[18]认为DSC>0.7表明勾画效果较好,而本研究所设计的自动分割模型的自动勾画效果优于此标准(DSC均大于0.89),说明所建模型具有较好的性能。
目前有很多关于OARs的自动分割的研究[19-22],其中大部分的研究都是关于头颈部OARs的自动分割。在乳腺癌的放射治疗过程中,相比其他胸部肿瘤,对健侧乳腺的保护也是不可缺少的。由于乳腺形态及类型的多样性,基于atlas的自动勾画技术的勾画准确性较差,在相同数量训练数据集的情况下,其对正常乳腺的勾画准确性较本研究所训练的基于U-Net的自动分割方法的自动分割准确性差。如图4所示,则可以看到在DSC折线图中,由基于U-Net的自动勾画模型勾画的健侧乳腺的DSC折线均位于由基于atlas的自动方法勾画的健侧乳腺的DSC折线之上。说明在每一例健侧乳腺的勾画中,基于U-Net的自动分割方法与手动勾画方法的勾画轮廓重合性均高于基于atlas的勾画结果(图3)。因此在对于健侧乳腺的自动勾画,本研究训练的方法优于基于atlas的自动勾画方法。本研究所训练的自动分割方法可以运用于临床并辅助医生对乳腺癌OARs的自动勾画,有利于减少医生的勾画时间,提高工作效率。本研究所训练的自动分割方法勾画的OARs适用于无锁骨以上靶区的乳腺癌放疗中周围正常组织的保护,而对于有锁骨以上靶区的乳腺癌则需要医师对另外所需的OARs进行补充勾画。因此在今后的研究中,可进一步增加臂丛、甲状腺、气管、脊髓等OARs作为训练对象,从而进一步减少医生补充修改勾画的时间。
4 结论
综上所述,基于U-Net的自动勾画模型在乳腺癌危及器官勾画中具有较好的勾画效果,且其勾画准确性较基于atlas的自动勾画方法的高。
[1] Deborah E,Citrin MD.Recent.developments in radiotherapy[J].N Engl J Med,2017,377(11):1065-1075.
[2] Han X,Hoogeman MS,Levendag PC,et al.Atlas-based autosegmentation of head and neck CT images[J].Med Image Comput Comput Assist Interv,2008,11(Pt2):434-441.
[3] Han X.WE-E-213CD-06:A locally adaptive,intensity-based label fusion method for multi- atlas auto-segmentation[J].Med Phys,2012,39(6Part27):3960.
[4] Dolz J,Laprie A,Ken S,et al.Supervised machine learningbased classification scheme to segment the brainstem on MRI in multicenter brain tumor treatment context[J].Int J Comput Assist Radiol Surg,2016,11(1):43-51.
[5] Tong N,Gou S,Yang S,et al.Fully automatic multi-organ segmentation for head and neck cancer radiotherapy using shape representation model constrained fully convolutional neural networks[J].Med Phys,2018,45(10):4558-4567.
[6] Zhu W,Huang Y,Tang H,et al.AnatomyNet: deep learning for fast and fully automated whole-volume segmentation of head and neck anatomy[J].Med Phys,2019,46(2):576-589.
[7] Schipaanboord B,Boukerroui D,Peressutti D,et al.Can atlasbased auto-segmentation ever be perfect?Insights from extreme value theory[J].IEEE Trans Med Imaging,2019,38(1):99-106.
[8] Taalimi A,Ensafi S,Qi H,et al.Medical image computing and computer-assisted intervention-MICCAI 2015[J].Lec Note Com Ence,2017,6361(1):69-87.
[9] Cardenas CE,Yang J,Anderson BM,et al.Advances in Auto-Segmentation[J].Semin Radiat Oncol,2019,29(3):185-197.
[10] Sun Y,Shi H,Zhang S,et al.Accurate and rapid CT image segmentation of the eyes and surrounding organs for precise radiotherapy[J].Med Phys,2019,46(5):2214-2222.
[11] Alom MZ,Yakopcic C,Hasan M,et al.Recurrent residual U-Net for medical image segmentation[J].J Med Imaging(Bellingham),2019,6(1):14006.
[12] Dong X,Lei Y,Wang T,et al.Automatic multiorgan segmentation in thorax CT images using U-net-GAN[J].Med Phys,2019,46(5):2157-2168.
[13] RTOG1106,“Randomized phase II trial of individualized adaptive radiotherapy using during-treatment FDG-PET/CT and modern technology in locally advanced non-small cell lung cancer (NSCLC)”[EB/OL].(2012-04-30)[2019-09-08].http://www.rtog.org/ClinicalTrials/ProtocolTable/StudyDetails.aspx?study=1106.
[14] Tsuji SY,Hwang A,Weinberg V,et al.Dosimetric evaluation of automatic segmentation for adaptive IMRT for head-and-neck cancer[J].Int J Radiat Oncol Biol Phys,2010,77(3):707-714.
[15] Teguh DN,Levendag PC,Voet PW,et al.Clinical validation of Atlas-based auto-segmentation of multiple target volumes and normal tissue structures in the head and neck[J].Int J Radiat Oncol Biol Phys,2011,81(4):950-957.
[16] Chalana V,Kim Y.A methodology for evaluation of boundary detection algorithmson medical images[J].IEEE Trans Med Imaging,1997,16:642-652.
[17] Poder J,Yuen J,Howie A,et al.Dose accumulation of multiple high dose rate prostate brachytherapy treatments in two commercially available image registration systemsr[J].Phys Med,2017,43:43-48.
[18] Zijdenbos AP,Dawant BM,Margolin RA,et al.Morphometric analysis of white matter lesions in MR images: method and validation[J].IEEE Trans Med Imaging,1994,13(4):716-724.
[19] Liang S,Tang F,Huang X,et al.Deep-learning-based detection and segmentation of organs at risk in nasopharyngeal carcinoma computed tomographic images for radiotherapy planning[J].Eur Radiol,2019,29(4):1961-1967.
[20] Men K,Dai J,Li Y.Automatic segmentation of the clinical target volume and organsat risk in the planning CT for rectal cancer using deep dilated convolutional neuralnetworks[J].Med Phys,2017,44:6377-6389.
[21] Ibragimov B,Xing L.Segmentation of organs-at-risks in head and neckCT images using convolutional neural networks[J].Med Phys,2017,44:547-557.
[22] Zhong T,Huang X,Tang F,et al.Boosting-based cascaded convolutional neural networks for the segmentation of CT organs-at-risk in nasopharyngeal carcinoma[J].Med Phys,2019,46(12):5602-5611.
Automatic Segmentation of Organs at Risk of Breast Cancer Using U-Net-Based Automatic Segmentation Method