PS/APP双转基因小鼠大脑中β-淀粉样蛋白的磁共振显微成像和组织切片研究
林岚,付振荣,张柏雯,周著黄,高宏建,吴水才
北京工业大学 生命科学与生物工程学院,北京 100124
[摘 要]目的探讨双转基因小鼠PS/APP大脑三维磁共振显微成像(MRM)的T2图与二维组织切片中Aβ斑块之间的关系。方法本文提出了一个高效、全自动的图像处理流程,即先通过未染色的二维组织切片重建三维脑模型,之后将其与三维MRM数据配准。配准是一个多步的过程,首先对邻接的二维切片进行配准,然后再进行迭代的二维和三维配准。结果该流程可以较好配准组织切片与MRM,并能在T2图和染色切片上发现一定的Aβ斑块间的对应关系。结论通过神经影像技术来对转基因小鼠大脑中的Aβ斑块进行监测,了解阿尔茨海默症(AD)中Aβ斑块的产生、沉积和清除过程。
[关键词]β-淀粉样蛋白;磁共振显微成像;阿尔茨海默病
0 引言
阿尔茨海默病(Alzheimer’s disease,AD)[1-2]是一种发病率随年龄增长而成倍增加的, 进行性发展的致死性神经退行性疾病,是痴呆的主要病因之一。临床表现为认知和记忆功能的不断衰退、日常生活能力进行性减退,并可能伴有各种神经精神症状和行为障碍。随着人类寿命的延长,AD正变得日益流行。在60岁以下只有1%的人群患有AD,而超过85岁的人中几乎50%都患有AD。AD的发病机制尚未精确地阐明,如遗传、免疫、环境及神经递质等都可能参与致病。所以AD病因病理具有多样性、异质性和复杂性等特点。AD的特征性病理改变为β淀粉样蛋白(β-amyloid Protein,A β)在大脑皮层和海马的沉积,从而形成老年斑、tau蛋白过度磷酸化形成的神经纤维缠结(Neurofibrillary Tangles,NFTs),并且会导致脑皮层和海马区神经元细胞的减少[3-4]。
典型老年斑的核心是由直径为5~10 nm的微丝组成的Aβ聚集而成。Aβ经神经元产生分泌后,会在脑内形成高度聚集的、可溶状态的寡聚体,然后进一步形成Aβ纤维而沉积在脑内。越来越多的研究结果显示,Aβ在AD的发生、发展过程中起着主导性的作用。例如, “淀粉样蛋白假说”[5]认为脑组织内Aβ的沉积是AD发生的最初起因,所有AD的其它病理改变,包括含有tau蛋白的NFTs形成、突触缺失和神经细胞死亡,都可能由于大脑内Aβ的清除速率小于其产生速率而引起的。治疗AD需要对不同阶段的Aβ进行不同的处理。在临床前期,目标为如何防止Aβ的产生;在轻度认知受损阶段,目标为怎样去除Aβ斑块;而在痴呆阶段,目标为设法解决Aβ的沉积。当前,科学研究的焦点主要集中于如何预防Aβ的形成以及怎样能抑制Aβ沉积而形成不可溶的斑块。
PS/APP双转基因的AD小鼠模型由于可以在小鼠大脑形成Aβ斑块,已被广泛运用于AD研究之中[6-7]。常用的实验方法是对小鼠大脑进行组织切片,一般通过固定、冰冻、数码照相等方法获取到组织切片。但这种方法工作量比较大,而且无法比较同一小鼠大脑随时间变化而产生的改变。当前,在动物模型的研究中,提供Aβ斑块的定量和可视化的神经影像技术变得越来越重要。磁共振显微成像(Magnetic Resonance Microscopy,MRM)已经被广泛用于小鼠模型上来对Aβ斑块进行检查[8-10]。在这项工作中,了解MRM成像中得到Aβ斑块与组织学切片间的关系是极为重要的。由于被研究的每一个组织切片的采集都是独立进行的,切片间的空间信息没有保存,所以这一工作在技术上还存在不少挑战。当前用的较多的方法是通过交互式软件实现手工配准[11]。一些自动化的方法虽然也有被提出,但在具体的实际操作中还是存在着复杂度较高的问题[12-13]。本研究的目标是优化自动化配准的方法,建立准确的三维组织学切片模型。对三维的组织学切片模型和MRM的T2图进行配准,获取Aβ斑块的初期比较。
1 材料与方法
1.1 研究对象与数据获取
在研究中,笔者使用了一只C57BL/6J小鼠作为对比鼠,一只PS/APP双转基因小鼠作为实验鼠。小鼠的年龄均为18个月,此时PS /APP小鼠大脑中会有着明显的Aβ斑块。Bruker Biospec 4.7T/40 cm小动物MRM成像仪(Bruker BioSpin,Ettlingen,Germany)被用于进行图像采集。首先通过径向快速自旋回波扫描序列获得128张连续T2加权像。扫描参数为:TR/TE =1000 ms/76.5 ms,层厚0.1 mm,体素大小= 0.1 mm×0.1 mm×0.1 mm,FOV为256 mm×256 mm。由于Aβ斑块会导致T2的弛豫时间的降低[14],需采集大脑的定量T2图[15]。T2图通过在k空间的中心使用特定的TE,而在k空间的外围使用其他TE数值生成。扫描参数为:TR=2000 ms,层厚0.5 mm,TE分别为40、64、 88、112 ms。体素大小= 0.1 mm×0.1 mm×0.5 mm, FOV为256 mm×256 mm。T2图通过将图像强度与4个有效TE进行单指数衰减拟合生成,直接反映了组织的弛豫时间。Aβ斑块会导致弛豫时间的降低,所以Aβ斑块在T2图中呈现为暗色斑点。
小鼠用10 mg/mL的戊巴比妥钠按40 mg/kg腹腔麻醉后,经左心室插入灌流针,先灌注冰冻无菌生理盐水再灌注4%多聚甲醛。灌注同时切开小部分肝脏,断头取脑,放入同等浓度多聚甲醛外固定24 h。随后,大脑将连续通过10%蔗糖6 h,20%蔗糖6 h,30%蔗糖6 h进行梯度脱水,最后在德国来卡2135型切片机中制成50 μm厚的冰冻切片。高分辨率尼康金相显微镜(Nikon Optiphot)和尼康数码相机被用于组织切片的拍照。在低倍率放大时,由于显微镜内在原因造成的光照的不均匀非常明显。因此,笔者先对没有组织标本的载玻片进行拍照,以获取光照掩模。反转的光照掩模被用于校正图像灰度。为了与MRM的中的Aβ斑块进行对比,对组织切片通过硫代黄素S(1:1000)进行了染色处理(三维的T2加权像、三维的T2图、二维的未染色的组织切片、二维的硫代黄素S染色的组织切片)。
1.2 图像处理
图像处理算法均在MATLAB环境下实现(MATLAB 2013a, MathWorks Inc., Natick, MA, USA)。对于MRM图像,先将图像由原始文件格式转换为NIFTI格式。非参数非均匀强度归一化方法通过优化参数设计去除了MRM中的偏差场[16-17]。脑模板和自适应阈值的混合方法[18]被用于去除非脑组织。
对于组织切片图像,首先将图像由TIFF文件格式转换为NIFTI格式。与MRM相比,光学显微镜连续切片所生成的图像分辨率要高得多,可以识别细微的Aβ斑块。然而,直接对二维组织切片的连续部分进行简单堆叠并不能产生平滑的图像。对于采集的组织切片数据,形变主要发生在冰冻切片过程中,包括全局平移和旋转、部分的局部拉升和压缩以及一些图像伪影等。50 μm的切片厚度相对于组织的形状改变可以忽略,采用下列步骤实现二维组织切片图像的三维重建:① 对相邻的二维组织切片采用标准化的互信息法进行仿射变换配准,消除全局形变,得到一个初步三维组织切片模型;② 采用标准化的互信息法配准三维组织切片模型与三维的MRM,得到组织切片与MRM的对应关系;③ 采用基于互信息的B样条非刚体配准在二维尺度配准MRM(参照图像)和切片图像(移动图像);④ 返回到第二步,进行多次迭代,直到最终结果收敛,保留相应形变场信息。
将染色的组织切片与未染色的组织切片进行刚体配准,运用形变场信息将染色的组织切片配准到统一空间。将三维的T2加权像、三维的T2图进行刚体配准,运用形变场信息将三维的T2图配准到统一空间。现在四种图像都被配准到统一空间,便于相互比较。
2 结果
在本研究采集的两只小鼠脑组织切片图像共计282张。原始图像的大小为1280×1024(像素大小8.1 μm)。为了节省计算空间,与MRM图像相比较,对其进行重采样,图像大小为320×256(像素大小32.5 μm)。整个处理过程是全自动的,在一台标准PC(酷睿i5 3450,8 G内存)上耗时约30 min (其中约一半的时间是用于消除MRM图像中的偏差场)。由于显微镜在透射光照射下对数字图像的曝光不均匀,通过反转的光照掩模可以消除这种不均匀(图1)。通过三维重建算法重建的组织切片三维模型见图2。三维模型可以去除大部分高频噪音引起的合成形变,能够形成比较光滑和容易辨认解剖结构,重建效果较好。
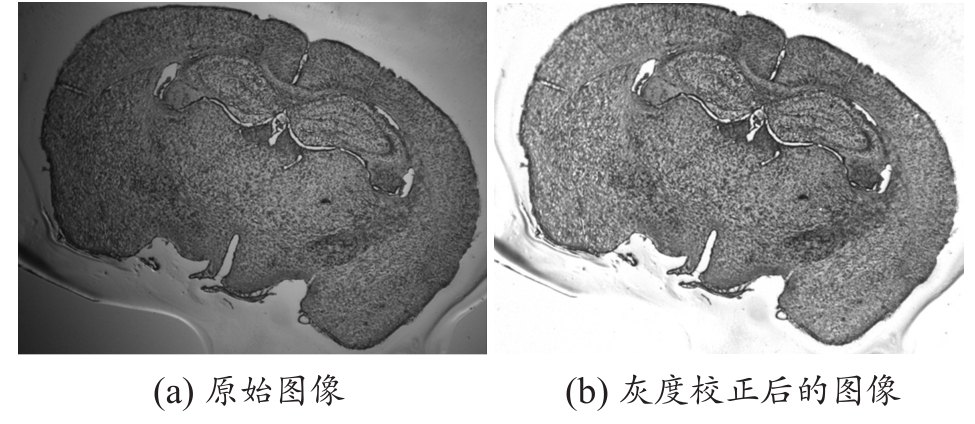
图1 组织切片灰度校正
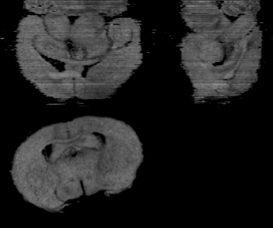
图2 三维重建的组织切片模型
为了比较模型的精度,采用Jaccard相似度来衡量三维组织切片模型与MRM图像的全脑体积的相似度。对于对比鼠,Jaccard相似度为0.972。而对于实验鼠,Jaccard相似度为0.968。同时也可通过手动配准建模来比较三维组织切片模型与MRM图像的全脑体积的相似度。对于手动配准建立的三维组织切片模型,Jaccard相似度为0.937和0.945。全自动建立的三维切片模型与MRM图像的全脑体积具有较高相似度。
三维染色组织切片模型与三维T2图配准后的结果显示,两者在Aβ斑块的检测上存在一定的对应关系。但不少染色组织切片上出现的Aβ斑块在T2图上没有相对应的暗点出现(图3)。这可能是由于组织切片的像素(8.3 μm)远远小于T2图的像素(100 μm)。很多Aβ斑块可能远远小于T2图体素的体积,所以无法进行检测。T2图更适合于检测较大的Aβ斑块。
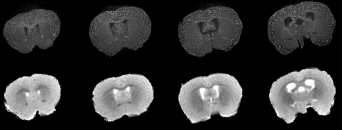
图3 配准后的染色组织切片与T2图
注:上图为硫代黄素S染色的组织切片,亮点为Aβ斑块;下图为T2图,暗点代表T2弛豫时间的降低,为可能的Aβ斑块。
3 讨论
本研究提出了一种建立三维组织切片模型的全自动方法,并将其与MRM三维图像进行配准。该方法能够较好地平衡配准精度和计算复杂度,但也存在一定的缺陷。例如,无论T2图还是组织切片,层间距较像素分辨率要低。考虑到进行三维插值会对Aβ斑块的检测造成影响,笔者提出切片是相对较均匀这一假设。配准算法主要用于建立相邻切片间的空间位置关系和修正二维切片内的形变,而对切片垂直方向的形变较为敏感。
4 结论
实验结果显示,尽管在染色组织切片与T2图之间不存在对应的Aβ斑块。但Aβ斑块在染色组织切片与T2图间却有一定的对应关系。这说明用3D组织学切片模型来检测小鼠大脑中的Aβ斑块的方法是可行的。未来本研究的重点是建立一个大型数据库,进一步研究MRM图像与组织学切片中Aβ斑块的关系。另外,相对于组织切片的Aβ斑块的研究,MRM的Aβ斑块检测还处于一个早期阶段。需要建立一种全自动算法,用来检测MRM的T2图中的Aβ斑块。这项研究可以加深对AD中Aβ斑块的产生、沉积和清除的了解。随着AD研究的重心正在逐渐转向AD的前期甚至无症状的早期诊断,通过神经影像技术来对转基因小鼠大脑中的Aβ斑块进行监测,可以帮助建立早期生物标志物,所以必将发展成为对抗AD的治疗方法。
[参考文献]
[1] Cahill S,Pierce M,Werner P,et al.A Systematic Review of the Public's Knowledge and Understanding of Alzheimer's Diseaseand Dementia[J].Alzheimer Dis Assoc Disord,2015,In Press.
[2] Hendrix J A,Finger B,Weiner MW,et al.The Worldwide Alzheimer's Disease Neuroimaging Initiative:An update[J].Alzheimers Dement,2015,11(7):850-859.
[3] Querfurth HW,LaFerla FM.Alzheimer's disease[J].N Engl J Med,2010,362(4):329-344.
[4] Ballard C,Gauthier S,Corbett A,et al.Alzheimer's disease[J].Lancet,2011,377(9770):1019-1031.
[5] Hardy J,Allsop D.Amyloid deposition as the central event in the aetiology of Alzheimer's disease[J].Trends Pharmacol Sci,1991,12(10):383–388.
[6] Yang D S,Kumar A,Stavrides P,et al.Neuronal apoptosis and autophagy cross talk in aging PS/APP mice,a model of Alzheimer's disease[J].Am J Pathol,2008,173(3):665-681.
[7] Holcomb L,Gordon MN,McGowan E,et al.Accelerated Alzheimertype phenotype in transgenic mice carrying both mutant amyloid precursor protein and presenilin 1 transgenes[J].Nat Med, 1998,4(1):97-100.
[8] Lin L,Fu Z,Xu X,et al.Mouse brain magnetic resonance microscopy:Applications in Alzheimer disease[J].Microsc Res Tech,2015,78(5):416-424.
[9] Jack CR Jr,Marjanska M,Wengenack TM,et al.Magnetic resonance imaging of Alzheimer's pathology in the brains of living transgenic mice:a new tool in Alzheimer's disease research[J].Neuroscientist,2007,13(1):38-48.
[10] Jack CR Jr,Garwood M,Wengenack TM,et al.In vivo visualization of Alzheimer's amyloid plaques by magnetic resonance imaging in transgenic mice without a contrast agent[J].MagnReson Med,2004,52(6):1263-1271.
[11] Rydmark M,Jansson T,Berthold CH,et al.Computer-assisted realignment of light microgragh images from consecutive section series of cat cerebral cortex[J].J Microsc,1992,165(Pt 1):29-47.
[12] Goubran M,de Ribaupierre S,Hammond R R,et al.Registration of in-vivo to ex-vivo MRI of surgically resected specimens: A pipeline for histology to in-vivo registration[J].J NeurosciMethods,2015,241:53-65.
[13] Ju T,Warren J,Carson J,et al.3D volume reconstruction of a mouse brain from histological sections using warp filtering[J].J NeurosciMethods,2006,156(1-2):84-100.
[14] Nabuurs RJ,Natté R,de Ronde FM,et al.MR microscopy of human amyloid-β deposits:characterization of parenchymal amyloid,diffuse plaques,and vascular amyloid[J].J Alzheimers Dis,2013,34(4):1037-1049.
[15] Altbach MI,Bilgin A,Li Z,et al.Processing of radial fast spinecho data for obtaining T2 estimates from a single k-space data set[J].Magn Reson Med,2005,54(3):549-559.
[16] Sled JG,Zijdenbos AP, Evans AC.A nonparametric method for automatic correction of intensity nonuniformity in MRI data[J].IEEE Trans Med Imaging,1998,17(1):87-97.
[17] Lin L,Wu S,Bin G.et al.Intensity inhomogeneity correction using N3 on mouse brain magnetic resonance microscopy[J].J Neuroimaging,2013,23(4): 502-507.
[18] Lin L,Wu S,Yang C.A template-based automatic skullstripping approach for mouse brain MR microscopy[J].Microsc Res Tech,2013,76(1):7-11.
Research of Magnetic Resonance Microscopy and Histological Section on Beta-Amyloid Plaques in PS/APP Transgenic Mice
LIN Lan, FU Zhen-rong, ZHANG Bai-wen, ZHOU Zhu-huang, GAO Hong-jian, WU Shui-cai
College of Life Science and Bio-engineering, Beijing University of Technology, Beijing 100124, China
Abstract:ObjectiveTo investigate the relationship between magnetic resonance microscopy (MRM) T2 map associated with Aβ plaques and their histology on dual transgenic PS/APP mouse brain.MethodsThe paper proposed an efficient, automated image-processing pipeline that used unstained sections to reconstruct a 3D brain model from 2D histological sections and further registered these with the corresponding 3D MRM. The imaging registration was a multistep registration procedure that first aligned the neighboring 2D slices and then iteratively performed 2D and 3D registration.ResultsThe registration pipeline can accurately register the 2D histology sections and 3D MRM. Some Aβ plaques can be found both in stained histology and MRM.ConclusionThis research monitored the Aβ plaques in PS/APP transgenic mice through neuroimaging technology, which can facilitate the understanding of the generation, deposition, and removal of Aβ plaques in Alzheimer’s disease.
[中图分类号]TP273
[文献标志码]A
doi:10.3969/j.issn.1674-1633.2016.02.007
[文章编号]1674-1633(2016)02-0031-03
收稿日期:2015-09-15
基金项目:北京市自然科学基金(7143171)资助。
通讯作者:林岚,副教授。
Abstract:: β-amyloid protein; magnetic resonance microscopy; Alzheimer’s disease